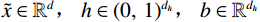Diffusion Models in Medical Imaging: A Comprehensive Survey
-
摘要:
以扩散模型为代表的生成式人工智能近年来在医学成像领域取得了迅猛的进展。为了帮助更多学者全面的了解扩散模型这一先进技术,本文旨在提供扩散模型在医学成像领域的详细概述。首先以扩散模型的起源演变为主线介绍扩散建模框架的基础理论和基本概念;其次根据扩散模型的特点提供其在医学成像领域的系统分类,并涵盖不同成像模态如磁共振成像、计算机断层扫描、正电子发射计算机断层显像和光声成像等的广泛应用;最后讨论目前扩散模型的局限性并展望未来研究的潜在发展方向,为研究者后续的探索提供了一个直观的起始点。部分代码开源在网址:https://github.com/yqx7150/Diffusion-Models-for-Medical-Imaging。
Abstract:Generative artificial intelligence represented by diffusion models has significantly contributed to medical imaging reconstruction. To help researchers comprehensively understand the rich content of diffusion models, this review provides a detailed overview of diffusion models used in medical imaging reconstruction. The theoretical foundation and fundamental concepts underlying the diffusion modeling framework were first introduced, describing their origin and evolution. Second, a systematic characteristic-based taxonomy of diffusion models used in medical imaging reconstruction is provided, broadly covering their application to imaging modalities, including magnetic resonance imaging (MRI), computed tomography (CT), positron emission computed tomography (PET), and photoacoustic imaging (PAI). Finally, we discuss the limitations of current diffusion models and anticipate potential directions of future research, providing an intuitive starting point for subsequent exploratory research. Related codes are available at GitHub: https://github.com/yqx7150/Diffusion-Models-for-Medical-Imaging.
-
神经网络驱动的生成式建模在深度学习领域迅速崛起并占据主导地位,其核心思想在于构造一个模拟数据生成过程的模型,即从已学习的数据分布中生成新样本。自生成模型兴起以来,其影响力已经广泛渗透到图像[1-2]、音频[3-4]转文本[5]和点云[6]等多个领域,推动了对复杂数据分布建模任务的巨大进展。从概率建模的角度来看,生成模型的核心特征在于其独特的训练方式,旨在确保生成的样本分布与原始数据的分布尽可能相似。为实现这一目标,传统的生成模型如能量模型(energy-based models,EBMs)采用在状态空间上定义非归一化概率密度的策略,通过调整模型参数来有效地学习样本数据分布。然而,这类模型在训练和采样期间需要进行计算量大且迭代缓慢的马尔可夫链蒙特卡罗采样[7]。
近年来,得益于深度学习的进步,人们重新拾起对生成模型的兴趣,产生了诸如生成对抗网络(generative adversarial networks,GANs)[8]、变分自编码器(variational autoencoders,VAEs)[9]和归一化流(normalizing flows,NFs)[10]等生成模型。这些模型在提高视觉保真度的同时加快了采样速度,但在实践中仍面临一些挑战,如训练时存在损失函数不稳定和后验分布估计偏移等问题。由此,基于扩散过程的生成模型(diffusion models,DMs)应运而生,为现有的EBMs、GANs、VAEs和NFs提供了一种替代方案,它们有效避免了匹配后验分布、估计配分函数以及引入额外网络约束等棘手问题。具体来讲,扩散模型包含了一个相互关联的双向过程:预定义的前向过程和相应的逆向过程。通常前向扩散过程将复杂的数据分布转化为简单的先验分布如高斯分布,而逆向扩散过程通过模拟常微分方程或随机微分方程[11-12]来逐步逆转前向过程重现样本数据。不同于上述生成模型,扩散模型采用固定的范式学习并且其隐变量具有与原始数据相同的高维度特性,因此为训练提供了更稳定的目标函数,同时在高质量样本生成方面取得了巨大的成功。表1从重建性能和运行效率等多方面展示了不同生成模型在医学成像任务中的优劣势。由此扩散模型开始受到广泛关注,生成式建模也逐步进入扩散模型时代。
表 1 五大生成模型全方位对比Table 1. Comprehensive comparison of five generative models特点/模型 VAE GAN EBM Flow Diffusion Model 基本原理 由编码器将数据映射到潜在空间概率分布,再由解码器生成数据 生成器和判别器对抗训练,生成器生成数据,判别器区分真假 定义一个可微的能量函数,将数据点的概率分布与其能量值联系 通过可逆变换将简单分布映射到复杂数据分布。 从噪声分布起,逐步去噪恢复目标数据 优点 1.生成质量高
2.训练稳定
3.潜在空间连续1.生成质量高
2.多样性好
3.应用广泛1.灵活性高
2.隐式生成
3.表示能力强1.高效样本生成和密度估计
2.可解释性强1.生成质量高
2.强大的建模能力
3.广泛的应用场景缺点 1.生成样本模糊
2.计算复杂度高
3.难捕捉复杂分布1.训练困难
2.对数据敏感
3.计算资源消耗大1.配分函数难计算
2.训练不稳定
3.采样效率低1.设计合适的变换
2.模块具有挑战性
3.计算资源需求高1.训练过程复杂
2.对噪声模型依赖性
3.生成速度较慢训练稳定性 稳定 不稳定 稳定 稳定 稳定 生成质量 较高 高 较高 高 高 计算资源需求 中等 高 中等 高 高 模型复杂度 中等 高 中等 高 高 可解释性 较好 较差 较好 好 较差 灵活性 较低 高 高 高 高 对数据分布假设 较强 较弱 较强 较强 较弱 数据质量敏感性 较低 高 较低 较低 较低 生成速度 中等 中等 中等 中等 较慢 随着深度学习技术的不断发展,扩散模型无论是在高层次的细节恢复还是多样性的示例生成方面,都展现出其令人印象深刻的能力。因此,扩散模型已被广泛应用于各大领域并凸显了其实用性,包括生成式建模任务如图像生成[13-22]、图像超分辨率重建[23-26]、图像修复[27-29],以及判别任务如图像分割[30-33]、分类[34]和异常检测[35-37]。特别地,扩散模型在医学成像领域的发展呈指数级增长,大量研究开始专注于探索扩散模型在医学成像领域中的应用。其中,将扩散模型作为生成先验应用于医学图像重建任务是该领域的一大研究热点。具体而言,医学图像重建的目标是从观测数据中推断原始数据的基本物理特性,常用方法通常依赖于网络模型的先验以指导重建过程。
扩散模型的独特之处在于因其使用真实的临床数据进行训练,所以能够学习数据中存在的复杂模式和结构以更准确地表示复杂数据分布[38-39],从而为重建过程提供更为准确的先验,以更精准的重建观测数据。由于常用传统方法难以对复杂数据建模,而扩散模型却适用于处理医学图像这类高维复杂数据,因此扩散模型能够提供一种更有效、更准确的方法来推断观测数据的基本物理特性,并能处理观测数据中的不确定性和噪声。
鉴于扩散模型在医学成像领域取得了显著的进步,对扩散模型进行系统性且模块化的分析显得尤为重要。近期,在计算机视觉领域涌现了大量关于扩散模型的综述[40-41]。然而,大多数这类综述并未将研究重点集中在医学成像领域的应用上。另一方面,目前大量国内外学者对于扩散模型在医学成像领域的研究仅聚焦特定的应用和图像模态。因此,关于扩散模型在医学图像重建应用的全面分析和系统性研究呈现出明显的知识空白,亟待进一步的探索和填补。所以,在医学成像领域对扩散模型进行全面而深入的分析和研究具备指导性意义。
本文调研大量基于扩散模型的医学图像重建论文和综述[42],进而对扩散模型在医学图像重建的发展(图1)和应用(图2)做出整体概述和全面总结,期望能为学者指引研究方向,为学术界的理论发展和实践应用提供有价值的参考和启示,从而为医学成像领域的进一步发展奠定坚实基础,有力推动该领域的创新与进步。
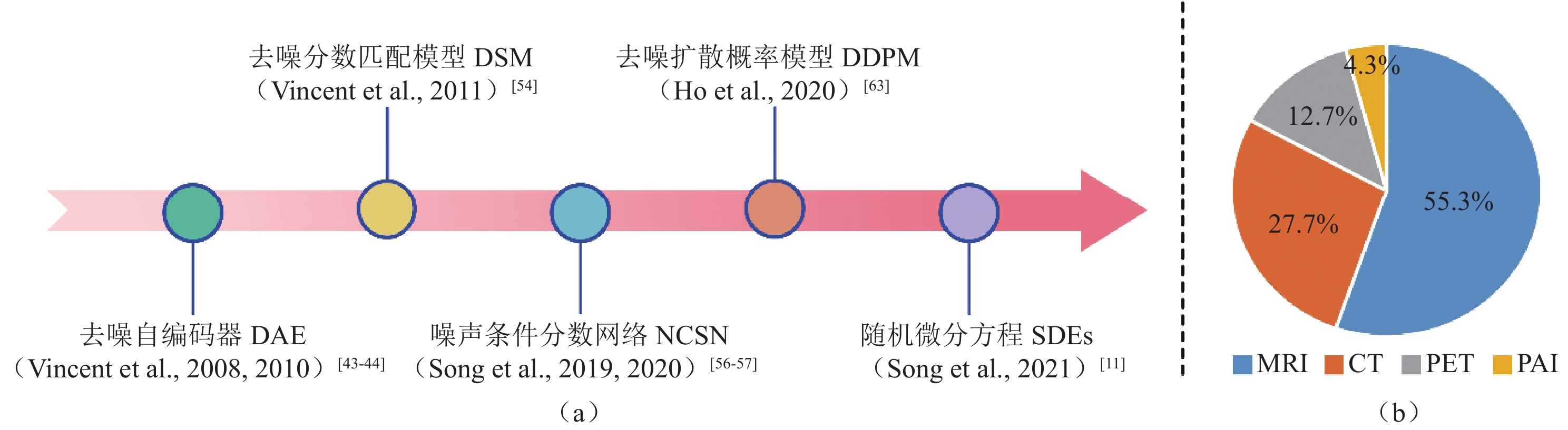
![]() 图 1 (a)扩散模型起源演变的时间轴;(b)按照关键词(diffusion model | medical imaging)、(score-based model | medical imaging)、(diffusion | medical | probabilistic model)在Google Scholar和Arxiv Sanity Preserver进行近5年相关论文检索,筛选并删除相同的结果最终统计扩散模型应用于不同成像模态分类的论文比例Figure 1. (a) Timeline of the origin and evolution of the diffusion model; (b) Relevant papers published within the last five years found via Google Scholar and Arxiv Sanity Preserver searches using the keywords (diffusion model | medical imaging), (score-based model | medical imaging), and (diffusion | medical | probabilistic model). Duplicate results were identified and deleted before calculating the proportion of papers applying a diffusion model to different imaging modality classifications
图 1 (a)扩散模型起源演变的时间轴;(b)按照关键词(diffusion model | medical imaging)、(score-based model | medical imaging)、(diffusion | medical | probabilistic model)在Google Scholar和Arxiv Sanity Preserver进行近5年相关论文检索,筛选并删除相同的结果最终统计扩散模型应用于不同成像模态分类的论文比例Figure 1. (a) Timeline of the origin and evolution of the diffusion model; (b) Relevant papers published within the last five years found via Google Scholar and Arxiv Sanity Preserver searches using the keywords (diffusion model | medical imaging), (score-based model | medical imaging), and (diffusion | medical | probabilistic model). Duplicate results were identified and deleted before calculating the proportion of papers applying a diffusion model to different imaging modality classifications首先,本文深入探究了扩散模型在医学图像重建应用中的起源和演变历程,并以此为基石详细地介绍了构成扩散模型框架的理论基础和核心概念。其次,本文根据扩散模型的独特性,系统性地梳理了其在医学图像重建的应用分类,该分类广泛涵盖了各种医学成像模态,如磁共振成像(magnetic resonance imaging,MRI)、计算机断层扫描(computed tomography,CT)、正电子发射计算机断层扫描(positron emission computed tomography,PET)、以及光声成像(photoacoustic imaging,PAI)等。最后,本文探讨了当前扩散模型存在的局限性,并前瞻性地展望了未来研究的潜在发展方向。例如,目前的研究趋势是将扩散模型与大模型相结合。
然而,由于医学数据的稀缺性,如何有效地利用小样本数据进行大模型的研究已成为未来探讨的关键议题之一,也为医学成像领域的创新和发展提供了新的研究方向和机遇。
本文主要贡献总结如下:
(1)本文是第1篇全面涵盖扩散模型在医学图像重建应用的中文综述。从扩散模型的起源和演变出发,系统性地梳理了扩散模型的基础理论和核心概念,旨在揭示其内在原理的发展轨迹;
(2)本文回顾扩散模型主要在医学图像重建应用的进展,包括MRI、CT、PET和PAI等多种成像模态。同时,模块化地分析并概述了针对不同成像模态的算法改进及创新性突破,意在全面展现扩散模型在医学图像重建的广泛应用和显著贡献;
(3)最后,本文探讨扩散模型在成像算法及实际应用中所面临的挑战和亟待解决的问题,并在此基础上明确了该领域的新兴发展趋势和潜在研究方向。
1. 扩散模型在医学成像中的应用——模型演变
扩散模型作为一种前沿的生成模型,在学习复杂数据分布方面已经证明了其高效性,并在多个应用领域展现了其广泛用途。尤其在医学成像领域,对扩散模型的研究兴趣持续升温。本节将以扩散模型的起源演变为基石,介绍扩散建模框架的基础理论和基本概念。
首先,深入探讨扩散模型的基础——去噪自编码器(denoising autoencoders,DAE)的相关理论。其次,详细地介绍基于去噪自编码器与分数匹配方法(score matching,SM)的去噪分数匹配模型(denoising score matching,DSM),该模型为后续扩散模型的发展提供了理论依据。接着,从两个主要视角进一步切入扩散模型的发展,分别是分数视角下的噪声条件分数网络(noise-conditioned score networks,NCSN)和变分视角下的去噪扩散概率模型(denoising diffusion probabilistic models,DDPM)。最后,概述了噪声条件分数网络和去噪扩散概率模型的连续化过程随机微分方程(stochastic differential equations,SDES)。
1.1 扩散模型的基石——去噪自编码器
作为经典自编码器神经网络的一种简单改进,去噪自编码器的训练目的并非仅限于将输入的样本进行重建,而是在输入的样本中人为引入噪声,并通过去噪还原方式解决各种复杂数据建模和特征学习的问题[43-49]。多项研究表明去噪自编码器能够有效地捕获输入数据中的特征,并通过去除所引入的多样性噪声来提高网络的泛化能力,从而广泛运用于不同图像处理场景[50-52]。在训练过程中,由于数据点来源于连续实值的分布,因此去噪自编码器使用具有平方重构损失的线性解码器是合理的。同时通过绑定编码器和解码器的权重,两者共享相同的线性变换参数,从而减少模型的参数数量。
去噪自编码器允许在输入数据中引入各向同性的随机噪声扰动,使网络更具鲁棒性,其框架的详细描述如下:
(1)输入数据
$x$ 被协方差为$ \sigma^2\mathbf{I} $ 的加性高斯噪声破坏,产生含噪输入$\tilde x = x + \zeta ,\;\zeta \sim {{N}}\left( {0,\;{\sigma ^2}{\mathbf{{I}}}} \right)$ ,其条件密度为$ q_{\sigma}\left(\tilde{x}|x\right)=\displaystyle\dfrac{1}{\left(2\pi\right)^{d/2}\sigma^d}\exp\left(-\frac{1}{2\sigma^2} \left\| \tilde{x}-{x} \right\| ^2\right) $ ;(2)含噪样本
$\tilde x$ 通过非线性仿射映射被编码成一个隐变量表示:${ h} = {\text{encode}}(\tilde x) = {\text{sigmoid}}\left( {{\boldsymbol{W}}\tilde x + {b}} \right)$ ,其中$ \tilde x \in {\mathbb{R}^d},\;{ h} \in {(0,\;1)^{{d_h}}},\;{b} \in {\mathbb{R}^{{d_h}}} $ ,W是一个${d_h} \times d$ 的矩阵;(3)将隐变量
${h}$ 通过仿射映射解码,重构原始数据:$ {x^r} = {{\rm{decode}}({h})} = {{\boldsymbol{W}}^{\mathrm{T}}}{h} + {c} $ ,其中$ {c} \in {\mathbb{R}^d} $ ;(4)优化参数
$\theta = \{ {\boldsymbol{W}},\;{b},\;{c}\} $ 使期望的平方重构误差${\left\| {{x^r} - x} \right\|^2}$ 最小,即去噪自编码器最小化目标函数为:$$ \begin{split} & J_{\mathrm{DAE}_{\sigma}}(\theta)= \\ & {E}_{q_{\sigma } \left({\tilde{x} },\; x\right)}\left(\left\|\boldsymbol{W}^{T}\left(\mathrm{sigmoid}(\boldsymbol{W} \tilde{x} + {b})\right)+ {c}-x\right\|^{2}\right)。\end{split} $$ (1) 上述去噪自编码器对输入样本加噪声后再去噪声的过程表明了扩散模型的发展借鉴了这一基本思想,并在其基础上进行了更深层次的创新和改进,使其适用于更广泛的应用领域。因此,可以说去噪自编码器是扩散模型演变发展的关键组成部分。迄今为止,去噪自编码器及其改进算法在医学图像重建中已经取得了不错的成果。Tezcan等[53]引入变分自编码器为描述数据密度先验的工具,为重建过程提供了更为准确的先验。Liu等[54]采用增强型的去噪自编码器作为MRI重建的显式先验,使用逆向传播自编码器误差来实现MRI重建,有效地提高了MRI重建效率。此外,Wang等[55]采用小波变换策略将样本从图像域转换到特征域作为去噪自编码器的输入,通过在小波域中提取先验提高了其表示能力,从而改善了图像的重建质量。
另一项新颖的方法[56]在采取多通道增强技术拓展数据维度的同时利用去噪自编码器挖掘其高维先验,提高了先验的表示效果和鲁棒性能。作为去噪自编码器的另一种拓展,深度均值漂移先验在医学成像领域也展现出其强大的重建能力。详细来说,均值漂移通过计算局部密度的梯度,逐步将数据点移动到密度更高的区域,从而实现数据点的分组和聚类。深度均值漂移先验则将均值漂移的思想融入深度学习模型,以捕捉图像的非局部特性(如重复纹理、结构等),以此增强网络对图像局部和全局特征的建模能力,同时提高对噪声的鲁棒性。Zhang等[57]利用数据密度先验的梯度进行重建,提出了多通道增强的深度均值漂移先验,为医学图像重建提供了新的思路和方法。
上述研究表明去噪自编码器技术在不断的改进,为后续扩散模型的发展奠定了基础。
1.2 去噪自编码器的演变——去噪分数匹配模型
去噪自编码器的目标是在样本中引入噪声,然后通过网络学习去除噪声的过程。分数匹配则是一种用于估计概率模型的方法,其目标是最小化模型生成数据分数和真实数据分数之间的差异。早期研究[45]已经明确了分数匹配方法与对比散度的关联,将其与去除方差无穷小的高斯噪声的最佳过程联系在一起。并进一步指出,使用分数匹配方法来训练高斯二元受限玻尔兹曼机等效于训练一个带有额外正则化项的去噪自编码器。鉴于去噪自编码器和分数匹配方法之间的内在联系,Vincent等[45]从一个全新的视角将去噪自编码器的训练重塑为一种基于正则化项的分数匹配形式,并提出了一种新的参数估计模型——去噪分数匹配模型。
分数匹配是一种用于估计数据概率密度
$p(x;\theta )$ 的方法,该方法在多元高斯和独立分量分析模型上得到了证明。在概率统计学中,很多概率密度函数可以写成如下的形式,如无向图中的概率密度,通过贝叶斯公式推导出的后验概率分布等:$$ p\left( {x;\theta } \right) = \frac{{\exp \left( { - E ( {x;\theta } )} \right)}}{{Z(\theta )}}\text{,} $$ (2) 式中
$E(x;\theta )$ 为能量函数,$Z(\theta )$ 为配分函数,分数则表示为数据概率密度的梯度$ \psi (x;\theta ) = {\nabla _x}\log p(x;\theta ) $ 。通常配分函数是难以估计甚至无法计算的,导致无法得到准确的概率密度,进而无法生成样本。而分数匹配方法的目的就是估计出概率密度的一个近似表示$q(\tilde x;\sigma )$ ,并用这个近似表示来代替原本的概率密度。然而当样本数据为高维复杂数据类型时,式(2)是难以计算的。受去噪自编码器原理的启发,即使用成对的样本数据$(x,\;\tilde x)$ 训练模型实现原始数据的有效编码和重建,新的解决方案去噪分数匹配模型定义如下:$$ {J_{DSMq}}(\theta ) = {{{\mathrm{E}}}_{{q_\sigma }(x,\;\tilde x)}}\left( {\frac{1}{2} \left \| \psi (\tilde x;\theta ) - {\nabla _{\tilde x}}\log {q_\sigma }(\tilde x|x)\right \| {^2}} \right) 。 $$ (3) 当添加的噪声足够小时,有
$ {\nabla _{\tilde x}}\log q\left( {\tilde x;\sigma } \right) \approx {\nabla _x}\log p\left( {x;\theta } \right) $ 成立。基于此理想情况,可用分数匹配方法结合去噪自编码器思想估计出$ q\left( {\tilde x;\sigma } \right) $ 的分数$ {\nabla _{\tilde x}}\log q\left( {\tilde x;\sigma } \right) $ ,并用它近似表示原数据分布$ p\left( {x;\theta } \right) $ 的分数。总而言之,去噪分数匹配模型继承了去噪自编码器对有噪声样本进行去噪的思想,从而提高模型的鲁棒性和泛化能力。同时,从分数匹配方法中借鉴了非标准化概率密度的分数函数的概念,使其在处理噪声和复杂数据分布方面独具优势,为模型训练和数据重建提供了更为灵活有效的手段。此外,Block等[58]详细讨论了去噪自编码器在数据分布中的学习能力,并进一步揭示了它们在特定情况下的等价性,即优化去噪自编码器损失等同于优化去噪分数匹配模型损失。上述围绕去噪分数匹配模型展开的一系列研究为扩散模型的发展做出了重要贡献,也为其后续研究提供了关键的思路和方案。
1.3 不同视角下的扩散模型——噪声条件分数网络和去噪扩散概率模型
在去噪自编码器和去噪分数匹配模型发展的理论奠定下,扩散模型成为了生成式学习领域的一个前沿拓展,其主要目标是估计与数据概率密度相关的梯度(即“分数”)并使用朗之万动力学从估计的数据概率密度中进行采样来生成新的样本。
在详细介绍扩散模型的工作原理之前,图3直观地展示了扩散模型的基本架构,包括前向过程(数据逐步添加噪声)和逆向过程(从噪声中重建原始数据)。该模型在各种应用领域都展现出了强大的生成能力,其中也包括医学成像领域。而通过不同视角下去进一步分析扩散模型,是对其后续发展的重要研究方向之一。
![]() 图 3 扩散模型分别在图像域和K空间域的基本框架。其中前向过程从初始数据开始逐步添加噪声,使其最终接近纯噪声分布;逆向过程通过学习逐步去噪,从随机噪声中还原为高质量的目标数据Figure 3. Basic framework of the diffusion model in the image and K-space domain. The forward process gradually adds noise from the initial data to approach a pure noise distribution; the reverse process gradually removes noise through learning and recovers high-quality target data from random noise
图 3 扩散模型分别在图像域和K空间域的基本框架。其中前向过程从初始数据开始逐步添加噪声,使其最终接近纯噪声分布;逆向过程通过学习逐步去噪,从随机噪声中还原为高质量的目标数据Figure 3. Basic framework of the diffusion model in the image and K-space domain. The forward process gradually adds noise from the initial data to approach a pure noise distribution; the reverse process gradually removes noise through learning and recovers high-quality target data from random noise1.3.1 分数视角下的扩散模型——噪声条件分数网络
分数视角下的扩散模型依赖于基于最大似然的估计方法,并使用数据概率密度
$p(x;\theta )$ 的分数函数来估计扩散过程的参数,噪声条件分数网络[46]就是其中的一个例子,即将去噪自编码器和去噪分数匹配模型的思想引入网络中作为梯度估计的方法。简而言之,噪声条件分数网络常用分数匹配方法估计不同噪声水平下受扰动数据的概率密度的梯度,然后通过朗之万动力学进行采样生成数据样本。朗之万动力学本质上是一种基于随机微分方程的物理模型,描述了粒子在势场中受到外力和随机热噪声作用下的运动。通过引入随机性和确定性相结合的动态过程,并结合梯度信息指导采样方向,朗之万动力学采样能够使得样本分布逐步收敛于目标分布,从而实现从复杂的概率分布中生成样本。通常数据概率密度
$p(x;\theta )$ 的分数函数定义为该数据概率密度的梯度$ {\nabla _x}\log {p_\theta }(x) $ ,而为了估计这个分数函数,可以训练一个带有分数函数的参数化神经网络$ {s}_{\theta }(\cdot)\approx {\nabla }_{x}\mathrm{log}{p}_{\theta }(x) $ 。具体来说,它的训练方式是通过最小化以下目标来近似$p(x;\theta )$ 的分数:$$ {{E}_{x \sim p(x)}}\left\| {{s_\theta }(x) - {\nabla _x}\log {p_\theta }(x)} \right\|_2^2 。 $$ (4) 然而,随着网络的层数增加,
$ {\nabla _x}\log {p_\theta }(x) $ 的计算难度也逐级递增。为了缓解这个问题,Song等[46]提出利用去噪分数匹配和切片分数匹配的方法[47]来训练网络。切片分数匹配的核心思想是通过随机投影将高维数据的分数函数投影到多个低维子空间(称为“切片”),然后在这些低维子空间上进行匹配。切片分数匹配是分数匹配方法的一种变体,通常针对高维数据分布建模的挑战进行了优化。相比而言,去噪分数匹配的计算速度较快而且能很好地估计受噪声干扰数据的概率密度的梯度,尤其适用于高维数据建模和生成建模领域,进而生成更高质量的图像。同时,由于实际的数据集中嵌入在高维空间的低维流形上,因此在低密度区域估计分数函数是不准确的。鉴于这一思考,Song等[47]引入多尺度的高斯噪声这一条件来扰动数据解决该问题。在他们的工作中,通过噪声分布$ {p_{{\sigma _t}}}\left( {{x_t}|x} \right) = {{N}}( {x_t}:x, {\sigma _t} \cdot {\boldsymbol{I}} ) $ 推导出${\nabla _x}\log \left( {{p_{{\sigma _t}}}( x )} \right)$ 可以等价于$ {\nabla _{{x_t}}}\log {p_{{\sigma _t}}}\left( {{x_t}|x} \right) = - {\displaystyle\frac{{{x_t} - x}}{{\sigma _t^2}}} $ ,其中,${x_t}$ 是$x$ 的有噪声状态。因此,对于给定的多尺度高斯噪声$ {\sigma _1}< {\sigma _2} < \cdots < {\sigma _T} $ ,噪声条件分数网络的目标函数可表示为:$$ \frac{1}{T}\sum\limits_{t = 1}^T {\lambda ({\sigma _t}){{E}_{p(x)}}{{E}_{{x_t} \sim {p_{{\sigma _t}}}({x_t}|x)}}\left\| {{s_\theta }\left( {{x_t},\;{\sigma _t}} \right) + \frac{{{x_t} - x}}{{\sigma _t^2}}} \right\|_2^2} \text{,} $$ (5) 式中
$\lambda ({\sigma _t})$ 为加权函数。采样过程是使用朗之万动力学的迭代过程完成的[59-60],朗之万动力学设计了一个马尔可夫链蒙特卡罗过程,仅使用分数函数$ {\nabla _x}\log {p_\theta }(x) $ 从概率密度$p\left( {x;\theta } \right)$ 中采样,迭代过程如下:$$ {x_t} = {x_{t - 1}} + \frac{\varepsilon }{2}{\nabla _{{x_{t - 1}}}}\log {p_\theta }\left( {{x_{t - 1}}} \right) + \sqrt \varepsilon \cdot {\omega _t} \text{,} $$ (6) 式中
${\omega _t} \sim {{N}}\left( {0,\;{\boldsymbol{I}}} \right),\;t \in \{ 1,\; 2,\;\cdots ,\;T\} $ 。当$\varepsilon \to 0$ 和$T \to \infty $ 时,由该过程生成的样本${x_t}$ 将收敛到初始样本的$ p\left( {x;\theta } \right) $ ,这一收敛过程称为退火朗之万动力学算法[46]。退火朗之万动力学结合了朗之万动力学的梯度驱动采样能力与退火策略的全局优化特性,通过动态调整噪声强度和平滑性,因此能够从复杂的概率分布中采样。总而言之,噪声条件分数网络是根据去噪自编码器和去噪分数匹配模型之间的一种新的联系而设计的,这一策略提高了整体的生成模型质量。受此思想启发,Quan等[61]进一步在噪声条件分数网络中使用多通道复制技术来提高样本生成效率,通过在高维空间中模拟多等级噪声,提出了同伦梯度生成先验算法。这一方法能够在高维空间中估计目标数据概率密度的梯度,有效避免了分数函数在低维流形和低数据密度区域不稳定的问题。如图4中的感兴趣区域残差图所示,噪声条件分数网络在图像细节恢复方面表现出明显优势,特别是在处理高频细节和复杂纹理结构时,优于传统重建算法和有监督深度学习算法。Zhu等[62]利用小波变换和自适应迭代策略的优点,通过在训练阶段形成小波张量作为网络输入,从而来训练噪声条件分数网络,实现了并行MRI的高精度重建。同样的思想也被He等[63]借鉴,他们将数据一致性作为条件项集成到低剂量CT重建。同时,通过对概率密度梯度进行退火处理,将模型的精细先验纳入迭代过程,实现了出色的去噪和重建效果。
综合上述基本原理和相关研究,噪声条件分数网络能够更精确地减少伪影和重建误差,同时保留原始图像的真实细节和纹理,这表明其在实现高保真度图像重建方面具有显著的潜力,为医学成像领域提供了可靠的解决方案。
1.3.2 变分视角下的扩散模型——去噪扩散概率模型
与噪声条件分数网络不同,变分视角下的扩散模型使用变分推断来近似目标概率密度,并通常采用最小化近似概率密度和目标概率密度之间的Kullback-Leibler散度的方式来优化模型。这类方法的典型代表是去噪扩散概率模型[13,48],它利用变分推断对扩散过程的参数进行估计,在有限时间内生成与原始数据分布匹配的高质量样本。具体而言,该模型设计了一个前向过程(将数据逐渐添加噪声)和一个逆向过程(通过去噪逐步还原数据),并通过对每一步的高斯分布参数化来约束生成过程,使得生成的样本具有高度的真实性和一致性。
由于变分推断方法能够有效地捕捉复杂分布,结合严格的数学框架和高效的生成机制,这类扩散模型展现出强大的生成能力,为高质量图像合成、重建以及其他生成任务提供了有力的工具支持。
前向的扩散过程。去噪扩散概率模型将前向扩散过程定义为一个马尔可夫链,该马尔科夫链具有当前状态仅依赖于前一个状态而与更早历史状态无关的特点。在这个马尔可夫链中,通过连续加入高斯噪声,得到一组含噪样本。考虑
$p({x_0})$ 为未加噪声的原始数据概率密度,给定数据样本${x_0}\sim p \left( {{x_0}} \right)$ ,前向加噪过程通过在时间$t$ 维度添加高斯噪声,从而能从${x_0}$ 得到${x_T}$ ,定义如下:$$ p\left( {{x_t}|{x_{t - 1}}} \right) = N\left( {{x_t}|\sqrt {1 - {\beta _t}} \cdot {x_{t - 1}},\;{\beta _t} \cdot {\boldsymbol{I}}} \right),\;\forall t \in \left\{ {1,\; \cdots ,\;T} \right\} \text{,} $$ (7) 式中
$T$ 和${\beta _1},\;{\beta _2},\; \cdots ,\;{\beta _T} \in \left[ {0,\;1} \right]$ 分别表示扩散步数和跨扩散步数的方差序列。${\boldsymbol{I}}$ 是单位矩阵,$N\left( x;\right. \left.\mu ,\;\sigma \right)$ 表示均值$\mu $ 和协方差$\sigma $ 的正态分布。考虑到$ {\alpha }_{t} = 1- {\beta }_{t} $ 和$ {\overline{\alpha }}_{t} = {{\displaystyle \prod }}_{s=0}^{t}{\alpha }_{s} $ ,所以可以直接对输入${x_0}$ 进行任意步长采样,如下所示:$$\begin{aligned} &p \left({x}_{t}|{x}_{0}\right)= N\left({x}_{t};\sqrt{{\overline{\alpha }}_{t}}{x}_{0},\;\left(1-{\overline{\alpha }}_{t}\right){\boldsymbol I}\right)\\& {x}_{t}=\sqrt{{\overline{\alpha }}_{t}}{x}_{0}+\sqrt{1-{\overline{\alpha }}_{t}}\grave{o} \end{aligned} 。$$ (8) 逆向的扩散过程。利用上述定义,可以近似地从
$p\left( {{x_0}} \right)$ 获得一个样本的逆向过程。鉴于此,直接从$ q\left({x}_{T}\right) = N\left({x}_{T};0,\;\boldsymbol{I}\right) $ 开始参数化该逆向过程如下:$$\begin{aligned} &{q_\theta }\left( {{x_{0:T}}} \right) = q\left( {{x_T}} \right)\prod\limits_{t = 1}^T {{q_\theta }\left( {{x_{t - 1}}|{x_t}} \right)} \\ & {q_\theta }\left( {{x_{t - 1}}|{x_t}} \right) = N\left( {{x_{t - 1}};{\mu _\theta }\left( {{x_t},\;t} \right),\;{\Sigma _\theta }\left( {{x_t},\;t} \right)} \right) \end{aligned}。$$ (9) 为了在训练时让
$ q({x_0}) $ 更好地逼近真实数据的概率密度$ p({x_0}) $ ,可以选择优化负对数似然的变分下界如下:$$ {{{E}}}\left[ { - \log {q_{0}}\left( {{x_{0}}} \right)} \right] \leq {{\text{\rm{B}}}_p}\left( { - \log \frac{{{q_{0}}\left( {{x_{{0}:T}}} \right)}}{{p\left( {{x_{1:T}}|{x_{0}}} \right)}}} \right)= {{{{E}}}_p}\left( { - \log q\left( {{x_T}} \right) - \sum\limits_{t \ge 1} {\log \frac{{{q_\theta }\left( {{x_{t - 1}}|{x_t}} \right)}}{{p\left( {{x_t}|{x_{t - 1}}} \right)}}} } \right) 。 $$ (10) Ho等[48]的研究发现,通过对扩散过程中的参数更新进行优化,并将其目标函数与噪声条件分数网络的目标函数相结合,所训练的模型权重能够更高效地进行图像重建,从而提升生成结果的精确性和一致性。此外,Lyu等[64]提出了一种条件去噪扩散概率模型,该模型通过对图像施加条件来控制扩散过程中的逆向步骤,实现了在特定任务上的定向生成。在MRI到CT的模态转换问题中,该方法取得了显著的成果,其优越性在多项指标上得到了验证(图5)。这些研究充分说明,去噪扩散概率模型在生成质量和评估指标方面优于其他生成式方法,不仅拓展了扩散模型的应用范围,也进一步凸显了其在医学成像领域中的卓越性能和发展潜力。
1.4 扩散模型的后发展——随机微分方程
噪声条件分数网络和去噪扩散概率模型都可视为离散化的概率模型,若将噪声尺度的数量推广到无穷大时,则定义了一个连续化过程——随机微分方程[11]。随机微分方程简单来说就是在传统常微分方程的基础上加入了随机扰动(通常为噪声项),因此该连续化过程能更加本质且精细的描述扩散过程,其形式如下:
$$ {\text{d}}x = f\left( {x,\;t} \right){\text{d}}t + g(t){\text{d}}w\text{,} $$ (11) 式中,
$ f(\cdot,\;t) $ 为随机微分方程的漂移系数,用于描述数据分布的期望变化;$g(t)$ 为扩散系数,用于控制噪声强度。$w$ 为标准布朗运动。设${x_0}$ 为未加噪的数据样本,${x_T}$ 近似标准高斯分布的扰动数据。从随机微分方程的角度来看,其目的就是要找到前向扩散过程关于时间的逆过程。而对于给定的前向过程,在一定条件下,存在逆方向扩散过程,该逆向过程定义为如下随机微分方程:$$ {\text{d}}x = \left( {f\left( {x,\;t} \right) - {g^2}(t){\nabla _x}\log {p_t}\left( x \right)} \right){\text{d}}t + g(t){\text{d}}\bar w \text{,} $$ (12) 其中
${\text{d}}t$ 是无穷小的反向时间步长,$\bar w$ 是逆向的布朗运动。由于逆向扩散过程从纯噪声图像${x_T}$ 起始,而真实数据的概率密度$p(x;\theta )$ 的梯度${\nabla _x}\log {p_\theta }(x)$ 是未知的,因此无法直接求出依赖真实数据概率密度的分数函数。为了求解其逆向过程,可以采用分数匹配方法训练模型来近似估计分数函数[11],其目标函数如下:$${ L}(\theta ) = {{ {E}}_{x(t)\sim q\left( {x(t)|x({0})} \right),\;x({0})\sim p(x)}} \Bigg( \frac{{\lambda (t)}}{2} \Big\| {s_\theta }\Big( {x(t),\;t} \Big)- {\nabla _{x(t)}}\log {q_t}\Big( {x(t)|x({0})} \Big) \Big\|_2^2 \Bigg)\; ,$$ (13) 其中,
$\lambda $ 为权重函数,$t \sim [{0},\;T]$ 为扩散时间。在基于分数的随机微分方程中,扩散过程首先逐步将真实数据转换为高斯噪声。随后,为了从高斯噪声中重新生成真实数据,在逆向扩散过程中引入了朗之万动力学采样。通过利用学习分数函数作为先验,该模型能够有效地建模数据分布在噪声干扰下的动态变化,并通过逆向过程实现高质量数据生成,尤其适用于医学图像重建任务。
基于此框架,Chung等[65]提出了一种针对MRI图像的去噪与超分辨率处理的多连续范式方法,利用随机微分方程算法来应对缺乏成对无噪声和含噪声数据的问题,从而实现了对低质量图像的精确恢复和增强。与此同时,为了解决多模态数据中常见的缺失问题,Meng等[66]提出了一种统一的基于多模态条件的分数生成模型。该模型将多余模态作为条件对缺失模态进行合成,并使用条件随机微分方程来估计不同模态的条件概率密度,效提升了多模态医学图像重建与合成的准确性。
综上所述,连续化扩散过程的引入为复杂图像重建和增强任务提供了强大的理论和技术支持。其灵活性和适应性使得扩散模型在医学图像处理中的潜力得以充分发挥,特别是在应对多模态数据融合、去噪、超分辨率以及缺失数据补全等挑战。
2. 扩散模型在医学成像中的应用——算法改进
如前文所述,扩散模型在医学成像领域的应用十分广泛,涵盖了多个成像模态,如MRI、CT、PET和PAI等。其出色的样本生成能力为医学研究和实践提供了更灵活、更鲁棒的方案,也为医学成像技术的发展和创新提供了全新的视角和契机。本节将以模块化的方式系统概述扩散模型在不同医学成像模态中的具体应用,并总结其在提升医学成像技术效率和重建精度方面的重要贡献。
2.1 磁共振成像
MRI是当今临床诊断和医学研究中重要的成像方法之一,但该方法采用频谱成像需要采集大量的K空间数据,因此成像时间较长,从而限制了其应用范围和使用效率。为了解决该问题,研究者们研究了各种重建技术力求在保证图像质量的前提下缩短数据采集时间。随着扩散模型在计算机视觉和图像处理等领域取得了重大突破,将其应用于磁共振成像重建也逐渐成为了研究热点[67-76]。Jalal等[38]首次将基于扩散生成模型构建的压缩感知重建框架应用于MRI,该工作采用基于分数匹配的生成模型进行训练,并成功实现了在不同采样条件下多线圈MR图像的重建。此项研究不仅在一个小型脑MRI数据集上展现出了优越的性能,而且为后验采样提供了理论支持。尤其值得注意的是,即使在临床数据面临严重分布偏移的情况下,该模型依然展现出卓越的适用性。此外,Song等[77]根据求解逆问题的前向模型的物理原理,在原有扩散模型的基础上进行改进,提高了模型的泛化能力使其适用于医学图像重建。该方法首先在医学图像上训练一个基于分数的生成模型以捕获样本数据先验分布,然后在重建阶段引入一种基于朗之万动力学采样的方法来生成目标对象。实验结果表明,该方法在没有充足的全采样图像数据的情况下依然能够实现较为精准的重建。
随后,Ye团队在扩散模型加速MR成像领域进行了大量研究,取得了显著成果[25,39,78]。例如,Chung等[39]通过去噪分数匹配网络来训练连续的与时间相关的分数函数。然后,在测试阶段使用数值随机微分方程求解器和数据一致性轮换迭代以实现重建。随着实验的进一步发现,扩散模型在采样速度上存在局限性,因此该团队进一步根据流形假设的思想提出了一个额外的校正项,通过减小样本数据分布和噪声分布之间的差异,实现更快收敛速率的同时减少了计算量[78]。大量的实验结果表明该团队的创新思想在迭代速率方面较先前方法更具优势,且适用于复杂应用场景。
不同于上述工作在低维图像空间进行扩散采样研究,Liu团队则将重点聚焦于MRI原始K空间域和高维空间域[79-83]。Tu等[79]引入加权策略和通道扩充技术将K空间数据映射到高维空间,从而训练了一个表示能力更强且泛化能力更鲁棒的噪声条件分数网络。同时,他们在重建阶段利用预测−校正器迭代采样,以此有效地生成与全采样数据相当的高质量图像。重建结果的定性与定量比较分别如表2和图6所示。从结果中能够清晰地观察到,在各类不同的采样方式下,SVD-WKGM 均展现出较高的峰值信噪比(PSNR)以及较低的重建残差。这表明该方法在重建效果方面有着卓越的泛化能力和较高的鲁棒性。
表 2 不同重建算法结果定量比较Table 2. Quantitative comparison between the results of different reconstruction algorithmsESPIRiT LINDBERG EBMRec SAKE WKGM SVD-WKGM T1 GE brain 2D random R=4 39.08/0.933 38.98/0.961 40.17/0.968 41.54/0.952 40.67/0.969 43.85/0.970 2D random R=6 36.01/0.921 35.16/0.958 36.55/0.952 38.09/0.932 37.14/0.957 39.94/0.960 T2 transverse brain 2D Poisson R=4 31.74/0.819 32.87/0.901 33.19/0.915 33.91/0.896 33.35/0.907 34.58/0.917 2D Poisson R=10 28.95/0.798 26.17/0.822 29.59/0.839 29.75/0.823 29.17/0.823 31.69/0.841 此外,该团队还提出了一种新颖的基于汉克尔矩阵的K空间扩散生成模型[80],其独特之处在于从仅包含单个K空间训练集中生成样本,为解决全采样数据样本难以获取的问题提供了新思路。具体来说,采用单个K空间数据构造成汉克尔矩阵并从中提取多个相互关联的补丁块,进而捕获不同补丁之间的分布情况使得扩散模型可以从冗余和低秩的数据空间中学习。实验结果表明单个K空间数据所构造的大量补丁块携带了足够的信息,使得扩散模型在小数据样本的情况下能够依据丰富的汉克尔先验重建出令人满意的结果。
受此启发,以Liang等为代表的团队着眼于传统扩散模型框架的深化和改进[84-87]并开展了一系列的工作。例如,他们通过贝叶斯深度学习从欠采样数据推断出全采样的数据分布,然后采用扩散模型前向过程扰动数据分布训练模型来估计其分布的梯度。同时,引入条件朗之万−马尔可夫链蒙特−卡罗采样提高模型的自适应性能[84]。图7显示了fastMRI膝关节数据的5倍随机欠采样和SIAT脑数据的4倍均匀采样的重建结果,结果表明该方法在混叠图案抑制和图像纹理细节恢复方面表现良好。Cui等[85]的研究中,将K空间中高频信息逐步衰减的过程建模为一个热扩散方程,通过这一框架形象地描述了高频信息在采样不足时逐渐丢失的物理机制。同时,从低频区域重建高频信息的逆过程则被表述为逆向热扩散方程,从理论上明确了恢复丢失信息的数学基础。进一步地,该工作结合热扩散方程的物理原理,引入磁共振成像(MRI)的物理模型对扩散过程进行了修正,使得模型能够更准确地模拟实际成像中的信号变化和噪声特性。在另一项研究中,该团队Cao等[86]深入挖掘扩散模型在图像重建任务中的鲁棒性,结合高频与低频信息分离的策略,创新性地设计了一种聚焦于高频空间扩散过程的新型模型。该模型通过将K空间中的高频分量与低频分量有效分离,优先处理对图像细节质量至关重要的高频信息,从而显著提升了图像的重建精度与细节还原能力。类似地,Guan等[87]进一步探索了扩散模型挖掘高频信息方法的优点,并提出了一种合理的原则以最大限度地联合利用这些方法实现高倍欠采样下MRI精准重建。本质上,该工作着眼于扩散过程中不同高频先验算子的融合以及相关多频先验信息的构造,从而约束扩散过程的噪声项更逼近于目标分布,实现精准重建的同时加速模型收敛。
这些研究共同推进了扩散模型在MR重建领域的发展,为未来进一步的研究提供了丰富的思路和方法。
2.2 计算机断层扫描
降低CT的剂量对于降低临床应用中的辐射风险至关重要,扩散模型的快速发展和广泛应用为CT成像重建算法的发展带来了新的方向[88-90]。代表性工作如Huang等[88]提出了一种创新方法,将投影数据转换为具有低秩特性的Hankel矩阵,并通过学习Hankel矩阵下的数据分布进行高效重建。在单样本训练的情况下,该方法能够达到相当于数千张样本训练的重建效果。此外,他们还将加权最小二乘法(PWLS)和全变分(TV)正则化嵌入扩散模型中,进一步提升了模型的重建质量。在此基础上,Zhang等[90]对该方法进行了改进,提出通过对投影数据进行角度分解,将其划分为多个角度区间,并对每个区间的数据进行多Hankel矩阵变换处理。通过扩散模型分别学习不同角度下的投影数据分布,该方法在单样本和少样本条件下显著提升了重建效果,进一步推动了稀疏数据训练的技术发展。
Liu等[91]将变换后的有限角正弦图应用于条件扩散模型从而获取更为丰富的数据先验,同时采用数据一致性模块改进算法以更准确地重建丢失信息。图8展示了人体CT描在60° 与90° 有限角度下的断层重建结果。与其他算法相比,该算法在细节恢复方面表现更为出色,PSNR值也处于较高水平。Xia团队的研究工作主要聚焦于去噪扩散概率模型的先验提取和后验采样过程[92-94]。通过在迭代重建框架中嵌入去噪扩散概率模型并引入Nesterov动量加速技术优化采样方式,改善了图像重建的质量和精度[92]。
Gao等[95]介绍了一种上下文误差调制的广义扩散模型,旨在解决低剂量CT图像去噪问题。该模型结合了上下文信息,根据误差模式调整扩散过程,有效地降低了噪声并提高了图像的质量。另一方面,Wu等[96]提出了一种将小波变换与扩散过程相结合的创新方法,该方法将小波子网络和扩散子网络融合于一个统一框架中,从而有效利用图像的多尺度信息和结构特征,因此在噪声抑制和图像质量提升方面取得了显著成效。
低剂量CT的重建结果如图9所示。此图对基于真实样本数据训练的模型,以及利用噪声样本训练的模型进行了对比,结果表明方法不仅能够有效去除噪声,保留更为丰富的纹理细节,同时还具备较高的PSNR值。Xu等[97]在此基础上采用小波变换分离投影域数据的高低频信息,并分阶段优化多个扩散模型。这种逐阶段的优化方法使得重建过程更为精细,可以更好地恢复丢失的图像信息。类似地,Zhang等[98]考虑到有限角度CT图像中伪影在不同频率范围内呈现出不同的分布,因此有必要在不同频率范围内对简化和特定的分布进行建模,以便更准确地估计概率密度并分离伪影,从而提高重建效果。因此,他们提出了一种波特启发的基础扩散模型,并采用小波变换将原始图像分解为多个频率分量,并在每个分量上分别应用扩散模型建模其概率密度。小波变换不仅能够保留空间对应关系,还能进行频率分解,保持伪影的方向特性,从而简化概率密度建模。
与上述研究不同,Guan等[99]提出了一种直接在投影域中进行扩散建模与采样优化的新方法。该方法充分利用投影域中的先验信息,通过在投影域内构建扩散模型来精确捕捉数据分布特性,并在采样优化过程中进一步增强重建效率。相比于传统图像域重建技术,这种直接从投影域出发的建模方式在保持数据一致性和提高重建质量方面展现出显著优势,为医学图像重建任务提供了新的技术路径和理论支持。
现有基于扩散模型的方法大多集中在单一域内进行处理,如图像域或正弦图域。虽然这些方法在一定程度上可以减少伪影,但它们往往无法充分利用跨域信息,限制了重建效果的提升。因此,Liu等[100]提出在正弦图域和图像域同时引入扩散先验,以恢复被金属伪影破坏的CT图像部分。通过引入双域策略,该方法不仅能够有效减少伪影,还能保持图像的细节和结构完整性,从而提供更高质量的重建结果。为了克服扩散模型计算成本高昂且无法捕捉细微结构的挑战,Wang等[101]提出了一种名为时间反转快速采样的新扩散模型结构。该新方法旨在通过引入时间反转机制、跳跃采样和压缩采样等技术,在保证高质量重建的同时显著减少所需的采样步骤,从而降低计算成本并提高重建效率。其核心在于如何在快速采样的同时保持图像的锐利边缘和精细特征,以实现更高效且高质量的稀疏角CT重建。除了在模型结构上,Wu等[102]从数据改造的角度出发,提出了一种多通道优化生成模型,旨在通过多通道融合充分利用数据一致性,并为生成模型提供更准确的指导,从而减少生成图像的随机性,提高重建的稳定性和准确性。这些创新性的研究为改善CT成像质量、减少噪声伪影和提高重建效率等提供了新的思路和方法,为CT在医学影像领域的精确诊断和治疗提供了新的机遇。
2.3 正电子发射计算机断层显像
PET技术作为目前核医学领域较为先进的成像技术,在早期诊断及治疗评估方面都有不俗的表现。但相对于MRI和CT,其成像原理决定了PET图像的分辨率不会太高,尤其是在限制注射示踪剂量与扫描时间的情况下。扩散模型因其对高维复杂数据的处理能力、生成高质量图像的能力以及对稀缺数据充分利用的能力,为PET图像重建领域提供了一种有前景的解决方案。
Shen团队在探索扩散模型在PET图像重建中的应用开展了一系列的工作[103-104]。例如,Han等[103]提出了一种由粗预测模块和细迭代模块相结合的新颖扩散框架,其中粗预测模块用来粗略地生成图像而细迭代模块用来对相应的残差图进行采样。此外该工作还额外引入了辅助引导策略和对比扩散策略并将其集成到重建过程中,在提高采样速度的同时改善了图像质量。Wang团队创新的将扩散模型应用于不同模态下PET图像联合重建应用[105-107]。以Xie等[105]的工作为例,通过引入不同模态数据如MRI作为引导并采用扩散模型进行联合概率密度估计,最终生成PET图像。此外,他们还提出了一种新型的联合扩散注意力模型[106]。该模型结合了联合概率分布和注意力机制,通过在扩散过程中向PET图像添加高斯噪声并保持引导图像MRI不变,实现了跨模态的生成。同时,通过采用相互一致性驱动的思想,进一步提出了新的PET-MRI联合重建策略[107],该策略利用扩散模型有效的整合两种成像模态的互补信息,在图像质量和处理速度方面都取得了显著的提升。Pan等[108]提出的基于扩散框架的PET一致性模型在改善低剂量PET图像质量方面表现出色,通过在逆向扩散过程中学习一致性函数,并利用平移窗口作为视觉变换器,以确保更佳的生成性能。
综上所述,这些创新性研究和方法为PET技术在减少辐射风险的同时保持高质量成像提供了新的思路。
2.4 光学和光声成像
PAI作为一种混合型非侵入式生物医学成像技术,结合了光学成像的高对比度和超声成像的高穿透深度特点。然而,传统重建在稀疏视角下可能导致光声图像质量低下。为应对这一问题,Song等[109]提出了一种基于扩散模型的稀疏重建方法。在该方法中,他们设计了一种基于分数的生成模型,用于学习数据分布的先验。并通过基于朗之万动力学采样的迭代重建,利用学习到的先验作为优化问题中数据一致项的约束,以获得最优解。重建结果如图10所示,结果表明该方法能从噪声中提取图像细节,重建血管细节。这一创新性的方法在稀疏视角下的光声断层成像中展现了更好的重建效果,不仅弥补了传统方法在稀疏视角下重建的不足,还充分利用了扩散先验,提高了图像重建的精度和准度。
类似地,Tong等[110]采用旋转一致性约束策略对扩散模型进行优化,通过朗之万动力学和数据一致项之间的迭代采样来恢复光声图像,成功地解决了从有限的探测数据中重建光声图像的难题。这种将物理成像原理与扩散模型相结合并融入迭代重建的方法为PAI技术的发展提供了新思路和解决途径,不仅在理论上具备重要意义,也在实际应用中具有重要价值。
3. 思考与展望
扩散模型作为一类强大的生成模型,在医学成像领域取得了显著进展,为诸如MRI、CT、PET和PAI等各类医学影像技术的改进提供了新的可能性。其应用不仅提高了图像的重建质量,还为临床诊断和医学研究提供了更精确、更可靠的数据支持。
然而,尽管扩散模型取得了显著的成就,仍需解决诸如生成过程速度较慢、对特定数据类型适用性受限以及无法有效降低维度等问题。这些问题或将成为未来研究方向的拓展点,需要不断探索,以进一步推动扩散模型在医学成像领域的发展与应用。本节将探讨扩散模型的缺陷并分析未来研究的大致方向,同时阐明在这个快速发展的领域继续研究的必要性。
医学数据稀缺是医学领域普遍面临的挑战之一。在医学成像领域,获取高质量、大规模的数据可能受到临床限制、隐私保护等因素的制约,因此如何利用小样本数据进行大模型研究将是未来的研究趋势之一。
在面对小样本数据的情况下,数据的构造和模型的设计变得至关重要,因为扩散模型需要更有效地利用有限的数据来提取并学习其中的关键特征,以避免过拟合从而提高泛化能力。在数据构造方面,通常的考虑是对原始数据进行分解变换并结合物理机制形成新的对象,从而增强小样本数据下多样性和有效性表示。这种数据构造方法有助于充分挖掘小样本数据的潜在信息,为扩散模型提供更全面、多维度的输入,提高了扩散模型对数据的敏感性和泛化能力,使其在小样本情境下更为可靠和稳健。
将Transformer框架[111]与扩散模型结合是未来研究的另一个重大方向。与卷积神经网络架构相比,Transformer框架具有更大的优势,包括能够对非本地交互进行建模并捕获数据中的长距离依赖关系。然而,尽管该框架有着巨大的潜力,但将其应用于扩散模型中仍处于探索阶段,需要进一步的研究来充分了解它们的能力和限制。
跨模态生成技术对医学成像领域具有重要意义,该技术的本质在于利用现有的数据生成其他模态的数据,从而能够提高数据丰富性和可用性,有助于提升诊断水平和促进医学研究的深入发展。然而,由于不同成像模态之间的数据分布和特征差异很大,且网络模型需要学习如何转换不同数据模态之间的复杂关系,因此克服这一挑战对模型而言需要大量的数据和复杂的网络结构设计。
扩散模型旨在通过学习潜在空间中有意义的隐变量来生成数据,同时具备强大的记忆能力和像素级别的视觉建模能力,所以在处理多模态数据时无需大量数据和复杂网络框架设计。例如,Wang等[112]提出了一种互信息引导扩散模型的无监督零样本学习方法,有效解决了零样本学习跨模态图像翻译的保真度问题。该模型通过利用不同模态之间的固有统计一致性学习将未见过的源图像翻译为目标模态。为了克服高维互信息计算的难题,进一步提出一种可微分的局部互信息先验,用于调节扩散模型的迭代去噪过程。通过捕获相同的跨模态特征作为隐变量,而不依赖于源和目标域之间的直接映射,使得该方法能够适应变化的源域,而无需重新训练,这在没有足够的标记源域数据时非常实用。因此,未来的研究可以继续探索如何更好地将不同模态的信息与扩散模型相融合,以拓展扩散模型在医学图像重建的应用。
为了降低不确定性和加速收敛,扩散模型通常避免在采样过程中选取范围较大的迭代步长,因为小范围的迭代步长有助于每一步生成的数据分布更接近真实数据分布。同时,当使用梯度下降来优化网络时,采用极大迭代步长可能将模型推向不确定的区域,导致训练困难。因此,为了提升采样效率和生成质量,扩散模型在设计采样策略时通常需要在采样速度与生成准确性之间权衡。Wei团队借助隐式神经表示指导扩散模型的后验采样,减少重建过程中不必要的采样步骤,有效平衡了采样效率和图像质量[113]。未来研究可以进一步探讨如何结合轻量化的网络结构与高效采样算法,降低计算复杂度的同时提升采样性能。例如,设计更加灵活的后验引导机制或结合其他高效优化方法,可能为扩散模型在高加速磁共振成像中的应用提供更优的解决方案。同时,如何使这些改进在有限资源环境下具有广泛适用性,也是一个具有研究价值的方向。
4. 结束语
本文围绕扩散模型在医学成像领域的应用进行了深入调研和全面分析,从多个维度系统性地总结了这一领域的研究进展与未来方向。通过回顾扩散模型的起源与演变,详细解析了其核心技术框架和理论基础,并重点梳理了其在不同成像模态下的实际应用。同时,本文结合当前研究热点,对扩散模型与大模型构建、跨模态生成等前沿技术的结合展开了深入讨论,提出了一些具有前瞻性的思考与建议,为学者在相关领域的后续研究提供了重要参考。
尽管本文强调了扩散模型在医学图像重建的快速发展及其潜在优势,但也认识到该领域尚处于早期探索阶段。许多技术挑战仍有待解决,包括高效模型设计、计算资源优化以及在临床场景中的实际应用等。
随着扩散模型在该领域的深入研究和广泛应用,希望本综述能够激发学者对扩散模型在医学成像领域的研究兴趣,并为未来在该领域的进一步探索起到启示作用。
-
图 1 (a)扩散模型起源演变的时间轴;(b)按照关键词(diffusion model | medical imaging)、(score-based model | medical imaging)、(diffusion | medical | probabilistic model)在Google Scholar和Arxiv Sanity Preserver进行近5年相关论文检索,筛选并删除相同的结果最终统计扩散模型应用于不同成像模态分类的论文比例
Figure 1. (a) Timeline of the origin and evolution of the diffusion model; (b) Relevant papers published within the last five years found via Google Scholar and Arxiv Sanity Preserver searches using the keywords (diffusion model | medical imaging), (score-based model | medical imaging), and (diffusion | medical | probabilistic model). Duplicate results were identified and deleted before calculating the proportion of papers applying a diffusion model to different imaging modality classifications
图 3 扩散模型分别在图像域和K空间域的基本框架。其中前向过程从初始数据开始逐步添加噪声,使其最终接近纯噪声分布;逆向过程通过学习逐步去噪,从随机噪声中还原为高质量的目标数据
Figure 3. Basic framework of the diffusion model in the image and K-space domain. The forward process gradually adds noise from the initial data to approach a pure noise distribution; the reverse process gradually removes noise through learning and recovers high-quality target data from random noise
表 1 五大生成模型全方位对比
Table 1 Comprehensive comparison of five generative models
特点/模型 VAE GAN EBM Flow Diffusion Model 基本原理 由编码器将数据映射到潜在空间概率分布,再由解码器生成数据 生成器和判别器对抗训练,生成器生成数据,判别器区分真假 定义一个可微的能量函数,将数据点的概率分布与其能量值联系 通过可逆变换将简单分布映射到复杂数据分布。 从噪声分布起,逐步去噪恢复目标数据 优点 1.生成质量高
2.训练稳定
3.潜在空间连续1.生成质量高
2.多样性好
3.应用广泛1.灵活性高
2.隐式生成
3.表示能力强1.高效样本生成和密度估计
2.可解释性强1.生成质量高
2.强大的建模能力
3.广泛的应用场景缺点 1.生成样本模糊
2.计算复杂度高
3.难捕捉复杂分布1.训练困难
2.对数据敏感
3.计算资源消耗大1.配分函数难计算
2.训练不稳定
3.采样效率低1.设计合适的变换
2.模块具有挑战性
3.计算资源需求高1.训练过程复杂
2.对噪声模型依赖性
3.生成速度较慢训练稳定性 稳定 不稳定 稳定 稳定 稳定 生成质量 较高 高 较高 高 高 计算资源需求 中等 高 中等 高 高 模型复杂度 中等 高 中等 高 高 可解释性 较好 较差 较好 好 较差 灵活性 较低 高 高 高 高 对数据分布假设 较强 较弱 较强 较强 较弱 数据质量敏感性 较低 高 较低 较低 较低 生成速度 中等 中等 中等 中等 较慢 表 2 不同重建算法结果定量比较
Table 2 Quantitative comparison between the results of different reconstruction algorithms
ESPIRiT LINDBERG EBMRec SAKE WKGM SVD-WKGM T1 GE brain 2D random R=4 39.08/0.933 38.98/0.961 40.17/0.968 41.54/0.952 40.67/0.969 43.85/0.970 2D random R=6 36.01/0.921 35.16/0.958 36.55/0.952 38.09/0.932 37.14/0.957 39.94/0.960 T2 transverse brain 2D Poisson R=4 31.74/0.819 32.87/0.901 33.19/0.915 33.91/0.896 33.35/0.907 34.58/0.917 2D Poisson R=10 28.95/0.798 26.17/0.822 29.59/0.839 29.75/0.823 29.17/0.823 31.69/0.841 -
[1] BAO J M, CHEN D, WEN F, et al. CVAE-GAN: Fine-grained image generation through asymmetric training[C]//Proceedings of the IEEE International Conference on Computer Vision, Venice, Italy, 2017: 2745-2754. DOI: 10.1109/iccv.2017.299.
[2] RAZAVI A, Van den OORD A, VINYALS O. Generating diverse high-fidelity images with VQ-VAE-2[J]. Advances in Neural Information Processing Systems, 2019, 32. DOI: 10.48550/arXiv.1906.00446.
[3] KONG Z F, PING W, HUANG J J, et al. Diffwave: A versatile diffusion model for audio synthesis[J]. arXiv Preprint arXiv: 2009.09761, 2020.
[4] OORD A, DIELEMAN S, ZEN H, et al. Wavenet: A generative model for raw audio[J]. arXiv Preprint arXiv: 1609.03499, 2016.
[5] LI X, THICKSTUN J, GULRAJANI I, et al. Diffusion-lm improves controllable text generation[J]. Advances in Neural Information Processing Systems, 2022, 35: 4328-4343. DOI: 10.48550/arXiv.2205.14217.
[6] YANG G D, HUANG X, HAO Z K, et al. Pointflow: 3D point cloud generation with continuous normalizing flows[C]//Proceedings of the IEEE/CVF international conference on computer vision, Seoul, Korea (South), 2019: 4541-4550. DOI: 10.48550/arXiv.1906.12320.
[7] BOND-TAYLOR S, LEACH A, LONG Y, et al. Deep generative modelling: A comparative review of VAEs, GANs, normalizing flows, energy-based and autoregressive models[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2021. DOI: 10.48550/arXiv.2103.04922.
[8] GOODFELLOW I, POUGET-ABADIE J, MIRZA M, et al. Generative adversarial nets[J]. Advances in Neural Information Processing Systems, 2014, 27. DOI: 10.48550/arXiv.1406.2661.
[9] JIMENEZ REZENDE D, MOHAMED S, WIERSTRA D. Stochastic backpropagation and approximate inference in deep generative models[J]. arXiv Preprint arXiv: 1401.4082, 2014.
[10] DINH L, SOHL-DICKSTEIN J, BENGIO S. Density estimation using real nvp[J]. arXiv Preprint arXiv: 1605.08803, 2016.
[11] SONG Y, SOHL-DICKSTEIN J, KINGMA D P, et al. Score-based generative modeling through stochastic differential equations[J]. arXiv Preprint arXiv: 2011.13456, 2020.
[12] KARRAS T, AITTALA M, AILA T, et al. Elucidating the design space of diffusion-based generative models[J]. Advances in Neural Information Processing Systems, 2022, 35: 26565-26577. DOI: 10.48550/arXiv.2206.00364.
[13] SOHL-DICKSTEIN J, WEISS E, MAHESWARANATHAN N, et al. Deep unsupervised learning using nonequilibrium thermodynamics[C]//International Conference on Machine Learning, Lille, France, 2015: 2256-2265. DOI: 10.48550/arXiv.1503.03585.
[14] SONG Y, DURKAN C, MURRAY I, et al. Maximum likelihood training of score-based diffusion models[J]. Advances in Neural Information Processing Systems, 2021, 34: 1415-1428. DOI: 10.48550/arXiv.2101.09258.
[15] SINHA A, SONG J M, MENG C L, et al. D2c: Diffusion-decoding models for few-shot conditional generation[J]. Advances in Neural Information Processing Systems, 2021, 34: 12533-12548. DOI: 10.48550/arXiv.2106.06819.
[16] VAHDAT A, KREIS K, KAUTZ J. Score-based generative modeling in latent space[J]. Advances in Neural Information Processing Systems, 2021, 34: 11287-11302. DOI: 10.48550/arXiv.2106.05931.
[17] SAHARIA C, HO J, CHAN W, et al. Image super-resolution via iterative refinement[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2022, 45(4): 4713-4726. DOI: 10.48550/arXiv.2104.07636.
[18] PANDEY K, MUKHERJEE A, RAI P, et al. Diffusevae: Efficient, controllable and high-fidelity generation from low-dimensional latents[J]. arXiv Preprint arXiv: 2201.00308, 2022.
[19] BAO F, LI C X, ZHU J, et al. Analytic-dpm: An analytic estimate of the optimal reverse variance in diffusion probabilistic models[J]. arXiv Preprint arXiv: 2201.06503, 2022.
[20] DOCKHORN T, VAHDAT A, KREIS K. Score-based generative modeling with critically-damped Langevin diffusion[J]. arXiv Preprint arXiv: 2112.07068, 2021.
[21] LIU N, LI S, DU Y L, et al. Compositional visual generation with composable diffusion models[C]//European Conference on Computer Vision, Tel-Aviv, Israel, 2022: 423-439. DOI: 10.48550/arXiv.2206.01714.
[22] JIANG Y M, YANG S, QIU H N, et al. Text2human: Text-driven controllable human image generation[J]. ACM Transactions on Graphics (TOG), 2022, 41(4): 1-11. DOI: 10.48550/arXiv.2205.15996.
[23] BATZOLIS G, STANCZUK J, SCHONLIEB C B, et al. Conditional image generation with score-based diffusion models[J]. arXiv Preprint arXiv: 2111.13606, 2021.
[24] DANIELS M, MAUNU T, HAND P. Score-based generative neural networks for large-scale optimal transport[J]. Advances in Neural Information Processing Systems, 2021, 34: 12955-12965. DOI: 10.48550/arXiv.2110.03237.
[25] CHUNG H, SIM B, YE J C. Come-closer-diffuse-faster: Accelerating conditional diffusion models for inverse problems through stochastic contraction[C]//Proceedings of the IEEE/CVF Conference on Computer Vision and Pattern Recognition, New Orleans, USA, 2022: 12413-12422. DOI: 10.48550/arXiv.2112.05146.
[26] KAWAR B, ELAD M, ERMON S, et al. Denoising diffusion restoration models[J]. Advances in Neural Information Processing Systems, 2022, 35: 23593-23606. DOI: 10.48550/arXiv.2201.11793.
[27] ESSER P, ROMBACH R, BLATTMANN A, et al. Imagebart: Bidirectional context with multinomial diffusion for autoregressive image synthesis[J]. Advances in Neural Information Processing Systems, 2021, 34: 3518-3532. DOI: 10.48550/arXiv.2108.08827.
[28] LUGMAYR A, DANELLJAN M, ROMERO A, et al. Repaint: Inpainting using denoising diffusion probabilistic models[C]//Proceedings of the IEEE/CVF Conference on Computer Vision and Pattern Recognition, New Orleans, USA, 2022: 11461-11471. DOI: 10.48550/arXiv.2201.09865.
[29] JING B, CORSO G, BERLINGHIERI R, et al. Subspace diffusion generative models[C]//European Conference on Computer Vision, Tel-Aviv, Israel, 2022: 274-289. DOI: 10.1007/978-3-031-20050-2_17.
[30] BARANCHUK D, RUBACHEV I, VOYNOV A, et al. Label-efficient semantic segmentation with diffusion models[J]. arXiv preprint arXiv: 2112.03126, 2021.
[31] GRAIKOS A, MALKIN N, JOJIC N, et al. Diffusion models as plug-and-play priors[J]. Advances in Neural Information Processing Systems, 2022, 35: 14715-14728. DOI: 10.48550/arXiv.2206.09012.
[32] WOLLEB J, SANDKÜHLER R, BIEDER F, et al. Diffusion models for implicit image segmentation ensembles[C]//International Conference on Medical Imaging with Deep Learning, New Orleans, USA, 2022: 1336-1348. DOI: 10.48550/arXiv.2112.03145.
[33] AMIT T, SHAHARBANY T, NACHMANI E, et al. Segdiff: Image segmentation with diffusion probabilistic models[J]. arXiv Preprint arXiv: 2112.00390, 2021.
[34] ZIMMERMANN R S, SCHOTT L, SONG Y, et al. Score-based generative classifiers[J]. arXiv Preprint arXiv: 2110.00473, 2021.
[35] PINAYA W H L, GRAHAM M S, GRAY R, et al. Fast unsupervised brain anomaly detection and segmentation with diffusion models[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention, New Orleans, USA, 2022: 705-714. DOI: 10.48550/arXiv.2206.03461.
[36] WOLLEB J, BIEDER F, SANDKUHLER R, et al. Diffusion models for medical anomaly detection[C]//International Conference on Medical image computing and computer-assisted intervention, New Orleans, USA, 2022: 35-45. DOI: 10.48550/arXiv.2203.04306.
[37] WYATT J, LEACH A, SCHMON S M, et al. Anoddpm: Anomaly detection with denoising diffusion probabilistic models using simplex noise[C]//Proceedings of the IEEE/CVF Conference on Computer Vision and Pattern Recognition, New Orleans, USA, 2022: 650-656. DOI: 10.1109/CVPRW56347.2022.00080.
[38] JALAL A, ARVINTE M, DARAS G, et al. Robust compressed sensing MRI with deep generative priors[J]. Advances in Neural Information Processing Systems, 2021, 34: 14938-14954. DOI: 10.48550/arXiv.2108.01368.
[39] CHUNG H, YE J C. Score-based diffusion models for accelerated MRI[J]. Medical Image Analysis, 2022, 80: 102479. DOI: 10.48550/arXiv.2110.05243.
[40] CAO H Q, TAN C, GAO Z Y, et al. A survey on generative diffusion model[J]. arXiv Preprint arXiv: 2209.02646, 2022.
[41] YANG L, ZHANG Z L, SONG Y, et al. Diffusion models: A comprehensive survey of methods and applications[J]. ACM Computing Surveys, 2023, 56(4): 1-39. DOI: 10.48550/arXiv.2209.00796.
[42] KAZEROUNI A, AGHDAM E K, HEIDARI M, et al. Diffusion models in medical imaging: A comprehensive survey[J]. Medical Image Analysis, 2023: 102846. DOI: 10.1016/jmedia.2023.102846.
[43] VINCENT P, LAROCHELLE H, BENGIO Y, et al. Extracting and composing robust features with denoising autoencoders[C]//Proceedings of the 25th international conference on Machine learning, Helsinki, Finland, 2008: 1096-1103. DOI: 10.1145/1390156.1390294.
[44] VINCENT P, LAROCHELLE H, LAJOIE I, et al. Stacked denoising autoencoders: Learning useful representations in a deep network with a local denoising criterion[J]. Journal of Machine Learning Research, 2010, 11(12). DOI: 10.5555/1756006.1953039.
[45] VINCENT P. A connection between score matching and denoising autoencoders[J]. Neural computation, 2011, 23(7): 1661-1674. DOI: 10.1162/NECO_a_00142.
[46] SONG Y, ERMON S. Generative modeling by estimating gradients of the data distribution[J]. Advances in Neural Information Processing Systems, 2019, 32. DOI: 10.48550/arXiv.1907.05600.
[47] SONG Y, GARG S, SHI J, et al. Sliced score matching: A scalable approach to density and score estimation[C]//Proceedings of The 35th Uncertainty in Artificial Intelligence Conference, Tel Aviv, Israel, 2020: 574-584. DOI: 10.48550/arXiv.1905.07088.
[48] HO J, JAIN A, ABBEEL P. Denoising diffusion probabilistic models[J]. Advances in Neural Information Processing Systems, 2020, 33: 6840-6851. DOI: 10.48550/arXiv.2006.11239.
[49] ERHAN D, COURVILLE A, BENGIO Y, et al. Why does unsupervised pre-training help deep learning?[C]//Proceedings of the 13th International Conference on Artificial Intelligence and Statistics, Pittsburgh, USA, 2010: 201-208.
[50] Le CUN Y, FOGELMAN-SOULIE F. Modeles connexionnistes de l’apprentissage[J]. Intellectica, 1987, 2(1): 114-143. DOI: 10.3406/intel.1987.1804.
[51] GALLINARI P, LECUN Y, THIRIA S, et al. Distributed associative memories: A comparison[M]. La Villette, Paris: Proceedings of Cognitiva 87, 1987.
[52] SEUNG H S. Learning continuous attractors in recurrent networks[J]. Advances in Neural Information Processing Systems, 1997, 10. DOI: 10.5555/3008904.3008997.
[53] TEZCAN K C, BAUMGARTNER C F, LUECHINGER R, et al. MR image reconstruction using deep density priors[J]. IEEE Transactions on Medical Imaging, 2018, 38(7): 1633-1642. DOI: 10.1109/TMI.2018.2887072.
[54] LIU Q G, YANG Q X, CHENG H T, et al. Highly under-sampled magnetic resonance imaging reconstruction using autoencoding priors[J]. Magnetic Resonance in Medicine, 2020, 83(1): 322-336. DOI: 10.1002/mrm.27921.
[55] WANG S Y, LV J J, HE Z N, et al. Denoising auto-encoding priors in undecimated wavelet domain for MR image reconstruction[J]. Neurocomputing, 2021, 437: 325-338. DOI: 10.1016/j.neucom.2020.09.086.
[56] LIU X S, ZHANG M H, LIU Q G, et al. Multi-contrast MR reconstruction with enhanced denoising autoencoder prior learning[C]//2020 IEEE 17th International Symposium on Biomedical Imaging, Iowa City, USA, 2020: 1-5. DOI: 10.1109/ISBI45749.2020.9098334.
[57] ZHANG M H, LI M T, ZHOU J J, et al. High-dimensional embedding network derived prior for compressive sensing MRI reconstruction[J]. Medical Image Analysis, 2020, 64: 101717. DOI: 10.1016/j.media.2020.101717.
[58] BLOCK A, MROUEH Y, RAKHLIN A. Generative modeling with denoising auto-encoders and Langevin sampling[J]. arXiv Preprint arXiv: 2002.00107, 2020.
[59] PARISI G. Correlation functions and computer simulations[J]. Nuclear Physics B, 1981, 180(3): 378-384. DOI: 10.1016/0550-3213(81)90056-0.
[60] GRENANDER U, MILLER M I. Representations of knowledge in complex systems[J]. Journal of the Royal Statistical Society: Series B (Methodological), 1994, 56(4): 549-581. DOI: 10.1111/j.2517-6161.1994.tb02000.x.
[61] QUAN C, ZHOU J J, ZHU Y Z, et al. Homotopic gradients of generative density priors for MR image reconstruction[J]. IEEE Transactions on Medical Imaging, 2021, 40(12): 3265-3278. DOI: 10.48550/arXiv.2008.06284.
[62] ZHU W Q, GUAN B, WANG S S, et al. Universal generative modeling for calibration-free parallel MR imaging[C]//2022 IEEE 19th International Symposium on Biomedical Imaging, Kolkata, India, 2022: 1-5. DOI: 10.1109/ISBI52829.2022.9761446.
[63] HE Z N, ZHANG Y K, GUAN Y, et al. Iterative reconstruction for low-dose CT using deep gradient priors of generative model[J]. IEEE Transactions on Radiation and Plasma Medical Sciences, 2022, 6(7): 741-754. DOI: 10.48550/arXiv.2009.12760.
[64] LYU Q, WANG G. Conversion between CT and MRI images using diffusion and score-matching models[J]. arXiv Preprint arXiv: 2209.12104, 2022.
[65] CHUNG H, LEE E S, YE J C. MR image denoising and super-resolution using regularized reverse diffusion[J]. IEEE Transactions on Medical Imaging, 2022, 42(4): 922-934. DOI: 10.48550/arXiv.2203.12621.
[66] MENG X X, GU Y N, PAN Y S, et al. A novel unified conditional score-based generative framework for multi-modal medical image completion[J]. arXiv Preprint arXiv: 2207.03430, 2022.
[67] LUO G, BLUMENTHAL M, HEIDE M, et al. Bayesian MRI reconstruction with joint uncertainty estimation using diffusion models[J]. Magnetic Resonance in Medicine, 2023, 90(1): 295-311. DOI: 10.1002/mrm.29624.
[68] PENG C, GUO P F, ZHOU S K, et al. Towards performant and reliable undersampled MR reconstruction via diffusion model sampling[C]// International Conference on Medical Image Computing and Computer-Assisted Intervention, Singapore, Singapore, 2022: 623-633. DOI: 10.1007/978-3-031-16446-0_59.
[69] GUNGOR A, DAR S U H, OZTURK S, et al. Adaptive diffusion priors for accelerated MRI reconstruction[J]. Medical Image Analysis, 2023: 102872. DOI: 10.1016/j.media.2023.102872.
[70] XIE Y T, LI Q Z. Measurement-conditioned denoising diffusion probabilistic model for under-sampled medical image reconstruction[J]. arXiv Preprint arXiv: 2203.03623, 2022.
[71] OZTURKLER B, LIU C, ECKART B, et al. SMRD: Sure-based robust MRI reconstruction with diffusion models[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention, Vancouver, Canada, 2023: 199-209. DOI: 10.1007/978-3-031-43898-1_20.
[72] MIRZA M U, DALMAZ O, BEDEL H A, et al. Learning fourier-constrained diffusion bridges for MRI reconstruction[J]. arXiv Preprint arXiv: 2308.01096, 2023.
[73] BIAN W Y, JANG A, LIU F. Diffusion modeling with domain-conditioned prior guidance for accelerated MRI and qMRI reconstruction[J]. arXiv Preprint arXiv: 2309.00783, 2023.
[74] KORKMAZ Y, CUKUR T, PATEL V M. Self-supervised MRI reconstruction with unrolled diffusion models[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention, Vancouver, Canada, 2023: 491-501. DOI: 10.1007/978-3-031-43999-5_47.
[75] CHEN L X, TIAN X Y, WU J J, et al. JSMoCo: Joint coil sensitivity and motion correction in parallel MRI with a self-calibrating score-based diffusion model[J]. arXiv Preprint arXiv: 2310.09625, 2023.
[76] RAVULA S, LEVAC B, JALAL A, et al. Optimizing sampling patterns for compressed sensing MRI with diffusion generative models[J]. arXiv Preprint arXiv: 2306.03284, 2023.
[77] SONG Y, SHEN L Y, XING L, et al. Solving inverse problems in medical imaging with score-based generative models[J]. arXiv Preprint arXiv: 2111.08005, 2021.
[78] CHUNG H, SIM B, RYU D, et al. Improving diffusion models for inverse problems using manifold constraints[J]. arXiv Preprint arXiv: 2206.00941, 2022.
[79] TU Z J, JIANG C, GUAN Y, et al. K-space and image domain collaborative energy-based model for parallel MRI reconstruction[J]. Magnetic Resonance Imaging, 2023, 99: 110-122. DOI: 10.1016/j.mri.2023.02.004.
[80] PENG H, JIANG C, CHENG J, et al. One-shot generative prior in hankel-k-space for parallel imaging reconstruction[J]. IEEE Transactions on Medical Imaging, 2023. DOI: 10.1109/TMI.2023.3288219.
[81] YU C M, GUAN Y, KE Z W, et al. Universal generative modeling in dual domains for dynamic MRI[J]. NMR in Biomedicine, 2023, 36(12): e5011. DOI: 10.1002/nbm.5011.
[82] GUAN Y, YU C M, LU S Y, et al. Correlated and multi-frequency diffusion modeling for highly under-sampled MRI reconstruction[J]. arXiv Preprint arXiv: 2309.00853, 2023.
[83] ZHANG W, XIAO Z W, TAO H, et al. Low-rank tensor assisted K-space generative model for parallel imaging reconstruction[J]. Magnetic Resonance Imaging, 2023, 103: 198-207. DOI: 10.1016/j.mri.2023.07.004.
[84] CUI Z X, CAO CT, LIU S N, et al. Self-score: Self-supervised learning on score-based models for MRI reconstruction[J]. arXiv Preprint arXiv: 2209.00835, 2022.
[85] CUI Z X, LIU C C, FAN X H, et al. Physics-informed deep MRI: K-space interpolation meets heat diffusion[J]. IEEE Transactions on Medical Imaging, 2024, 43(10): 3503-3520. DOI: 10.1109/TMI.2024.3462988.
[86] CAO C T, CUI Z X, LIU S N, et al. High-frequency space diffusion models for accelerated MRI[J]. arXiv Preprint arXiv: 2208.05481, 2022.
[87] GUAN Y, YU C M, CUI Z X, et al. Correlated and multi-frequency diffusion modeling for highly under-sampled MRI reconstruction[J]. IEEE Transactions on Medical Imaging, 2024, 43(10): 3490-3502. DOI: 10.1109/TMI.2024.3381610.
[88] HUANG B, LU S Y, LIU Q G, et al. One-sample diffusion modeling in projection domain for low-dose CT imaging[J]. IEEE Transactions on Radiation and Plasma Medical Sciences, 2024. DOI: 10.1109/TRPMS.2024.3392248.
[89] WU W W, WANG Y Y. Data-iterative optimization score model for stable ultra-sparse-view CT reconstruction[J]. arXiv Preprint arXiv: 2308.14437, 2023.
[90] ZHANG W H, HUANG B, LIU Q G, et al. Low-rank angular prior guided multi-diffusion model for few-shot low-dose CT reconstruction[J]. IEEE Transactions on Radiation and Plasma Medical Sciences, 2024. DOI: 10.1109/TCI.2024.3503366.
[91] LIU J M, ANIRUDH R, THIAGARAJAN J J, et al. DOLCE: A model-based probabilistic diffusion framework for limited-angle CT reconstruction[C]//Proceedings of the IEEE/CVF International Conference on Computer Vision, Paris, France, 2023: 10498-10508. DOI: 10.48550/arXiv.2211.12340.
[92] XIA W J, SHI Y Y, NIU C, et al. Diffusion prior regularized iterative reconstruction for low-dose CT[J]. arXiv Preprint arXiv: 2310.06949, 2023.
[93] XIA W J, CONG W X, WANG G. Patch-based denoising diffusion probabilistic model for sparse-view CT reconstruction[J]. arXiv Preprint arXiv: 2211.10388, 2022.
[94] XIA W J, NIU C, CONG W X, et al. Sub-volume-based denoising diffusion probabilistic model for cone-beam CT reconstruction from incomplete data[J]. arXiv e-Prints, 2023: arXiv: 2303.12861.
[95] GAO Q, LI Z L, ZHANG J P, et al. CoreDiff: Contextual error-modulated generalized diffusion model for low-dose CT denoising and generalization[J]. arXiv Preprint arXiv: 2304.01814, 2023.
[96] WU W W, WANG Y Y, LIU Q G, et al. Wavelet-improved score-based generative model for medical imaging[J]. IEEE Transactions on Medical Imaging, 2023. DOI: 10.1109/TMI.2023.3325824.
[97] XU K, LU S Y, HUANG B, et al. Stage-by-stage wavelet optimization refinement diffusion model for sparse-view CT reconstruction[J]. arXiv Preprint arXiv: 2308.15942, 2023.
[98] ZHANG J J, MAO H Y, WANG X R, et al. Wavelet-inspired multi-channel score-based model for limited-angle CT reconstruction[J]. IEEE Transactions on Medical Imaging, 2024, 43(10): 3436-3448. DOI: 10.1109/TMI.2024.3367167.
[99] GUAN B, YANG C L, ZHANG L, et al. Generative modeling in sinogram domain for sparse-view CT reconstruction[J]. IEEE Transactions on Radiation and Plasma Medical Sciences, 2023. DOI: 10.1109/TRPMS.2023.3309474.
[100] LIU X, XIE Y Q, DIAO S H, et al. Unsupervised CT metal artifact reduction by plugging diffusion priors in dual domains[J]. IEEE Transactions on Medical Imaging, 2024, 43(10): 3533-3545. DOI: 10.1109/TMI.2024.3351201.
[101] WANG Y Y, LI Z R, WU W W. Time-reversion fast-sampling score-based model for limited-angle CT reconstruction[J]. IEEE Transactions on Medical Imaging, 2024, 43(10): 3449-3460. DOI: 10.1109/TMI.2024.3418838.
[102] WU W W, PAN J Y, WANG Y Y, et al. Multi-channel optimization generative model for stable ultra-sparse-view CT reconstruction[J]. IEEE Transactions on Medical Imaging, 2024, 43(10): 3416-3475. DOI: 10.1109/TMI.2024.3376414.
[103] HAN Z Y, WANG Y H, ZHOU L P, et al. Contrastive diffusion model with auxiliary guidance for coarse-to-fine PET reconstruction[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention, Vancouver, Canada, 2023: 239-249. DOI: 10.1007/978-3-031-43999-5_23.
[104] JIANG C W, PAN Y S, LIU M X, et al. PET-diffusion: Unsupervised PET enhancement based on the latent diffusion model[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention, Vancouver, Canada, 2023: 3-12. DOI: 10.1007/978-3-031-43907-0_1.
[105] XIE T F, CAO C T, CUI Z X, et al. Brain pet synthesis from MRI using joint probability distribution of diffusion model at ultrahigh fields[J]. arXiv Preprint arXiv: 2211.08901, 2022.
[106] XIE T F, CAO C R, CUI Z X, et al. Synthesizing PET images from high-field and ultra-high-field MR images using joint diffusion attention model[J]. arXiv Preprint arXiv: 2305.03901, 2023.
[107] XIE T F, CUI Z X, LUO C, et al. Joint diffusion: Mutual consistency-driven diffusion model for PET-MRI co-reconstruction[J]. arXiv Preprint arXiv: 2311.14473, 2023.
[108] PAN S Y, ABOUEI E, PENG J B, et al. Full-dose PET synthesis from low-dose PET using high-efficiency diffusion denoising probabilistic model[J]. arXiv Preprint arXiv: 2308.13072, 2023.
[109] SONG X L, WANG G J, ZHONG W H, et al. Sparse-view reconstruction for photoacoustic tomography com-bining diffusion model with model-based iteration[J]. Photoacoustics, 2023, 33: 100558. DOI: 10.1016/j.pacs.2023.100558.
[110] TONG S Q, LAN H R, NIE L M, et al. Score-based generative models for photoacoustic image reconstruction with rotation consistency constraints[J]. arXiv Preprint arXiv: 2306.13843, 2023.
[111] DOSOVITSKIY A, BEYER L, KOLESNIKOV A, et al. An image is worth 16x16 words: Transformers for image recognition at scale[J]. arXiv Preprint arXiv: 2010.11929, 2020.
[112] WANG Z H, YANG Y Y, CHEN Y Z, et al. Mutual information guided diffusion for zero-shot cross-modality medical image translation[J]. IEEE Transactions on Medical Imaging, 2024, 43(8): 2825-2838. DOI: 10.1109/TMI.2024.3382043.
[113] CHU J Y, DU C H, LIN X Y, et al. Highly accelerated MRI via implicit neural representation guided posterior sampling of diffusion models[J]. Medical Image Analysis, 2025, 100.



 下载:
下载: